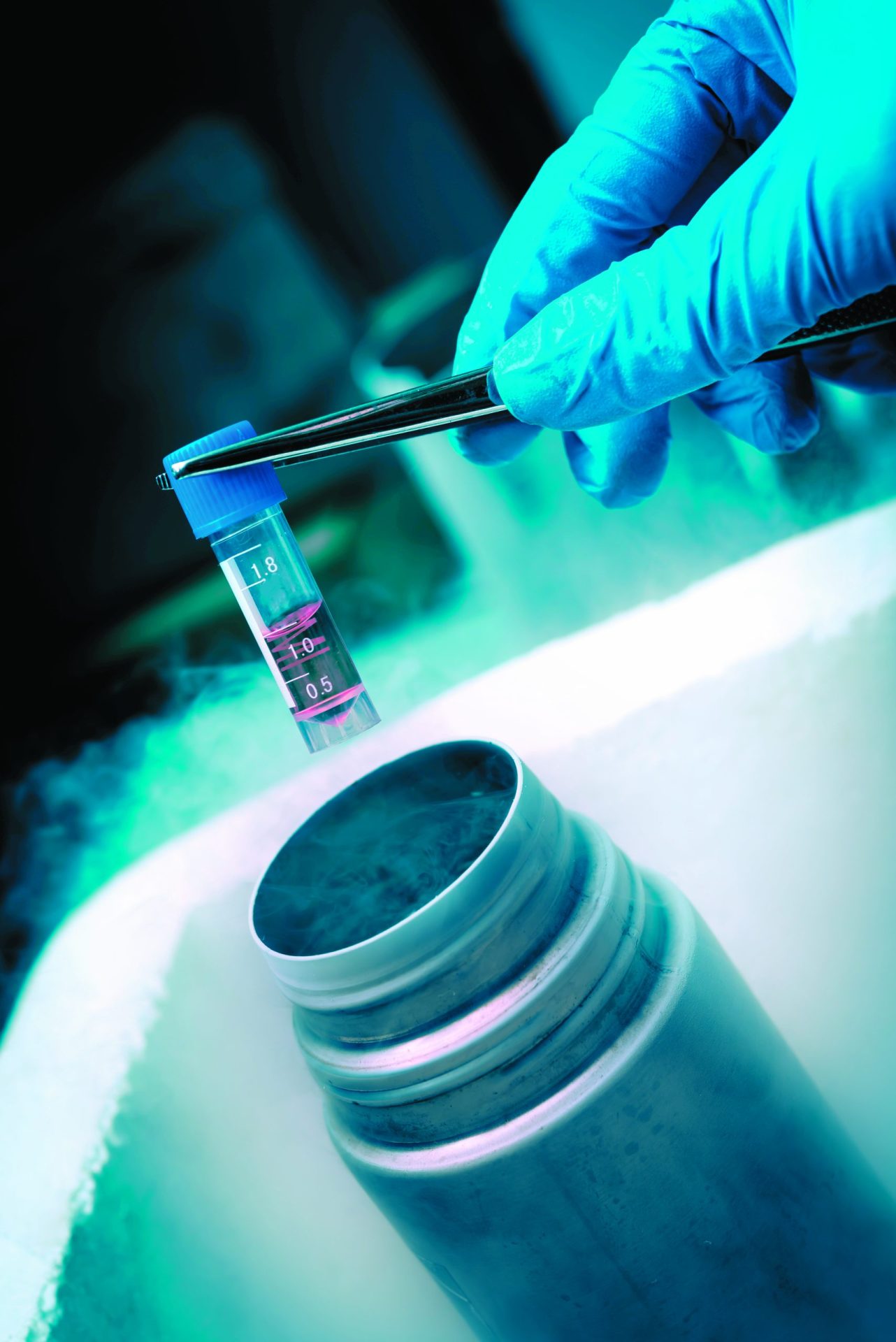
Imagine guardar todas as fotografias de família num tubo de ensaio, mais concretamente em pequenas partículas que nem lhe ocupariam a ponta de um dedo e com a garantia de que, daqui a milhares de anos, as gerações vindouras teriam acesso às imagens sem perda de qualidade.
Se é daqueles que nem descarrega as fotos do telemóvel, talvez não esteja a ver a utilidade desta ultra compactação de dados. Mas pense numa escala maior: nos milhões de fotografias guardadas na internet – as últimas estimativas sugerem que, a cada dois minutos, tiramos mais fotografias do que as que existiram ao longo de século e meio. Se lhe dissermos que, no futuro, alguns contentores com tubos de ensaio poderão substituir os servidores de gigantes como a Google ou a Amazon, vai querer saber como. A resposta está no ADN (sim, o ácido que codifica as instruções genéticas que fazem de nós o que somos).
Da medicina para o digital Se nas últimas décadas a genética era apontada como o futuro da medicina personalizada, há mais uma indústria que promete não ficar indiferente aos mistérios das sequências de letrinhas que codificam genes e às vezes, quando estão fora do sítio ou simplesmente faltaram à chamada, estão por detrás de doenças ou predisposições.
Os investigadores descobriram uma forma de usar a arquitetura do ácido desoxirribonucleico para armazenar informação, transformando os bits nestas mesmas letras – ou bases – do ADN: os nucleotídeos A, C, G e T.
Se o empreendimento não é inédito, na semana passada uma dupla de investigadores dos Estados Unidos anunciou nas páginas da revista “Science” ter desenvolvido um algoritmo que permite guardar 60% mais informação do que até aqui e recuperá-la sem comprometer os “ficheiros”, o que em experiências prévias tinha sido um obstáculo.
O trabalho, assinado por Yaniv Erlich e Dina Zielinski, cientistas computacionais da Universidade Columbia e do Centro de Genoma de Nova Iorque, foi desenvolvido em tempo recorde. Começaram em maio e apresentaram o artigo para publicação na “Science” em setembro do ano passado.
Dina Zielinski explicou ao i o processo. Para guardar informação em moléculas de ADN, começam por comprimir os ficheiros, convertendo-os em código binário: 0s e 1s.
O segredo da “eficácia” do processo está no algoritmo que desenvolveram, a que chamaram código “Fountain” (Fonte), e que permite codificar a informação de uma forma menos corruptível, partindo os dados em pequenos segmentos aleatórios e inserindo alguns 0s e 1s extra que permitem catalogar as partes do puzzle de forma a que, quando for hora de o converter de novo para imagem, áudio ou texto, esse processo ser mais fácil. Posto isto, segue-se a transcrição dos 0s e 1s para as quatro bases do ADN: 00 passa a ser o A do nucleótide adenina; 01 o T de timina; 10 converte para o C de citosina e 11 para o G de guanina.
Com a receita “genética” do ficheiro em mãos, os investigadores enviaram depois as sequências para uma biotecnológica, que produz ADN sintético. Neste caso, em vez de conter instruções para a vida, guardava, alguns registos singulares.
A equipa quis “divertir-se” um pouco, como explicaram à imprensa internacional, e escolheram armazenar no ADN um sistema operativo completo, um vírus informático, um texto do pai da teoria da informação Claude Shannon e um dos primeiros filmes dos irmãos Lumière, “L’Arrivée d’un train en gare de La Ciotat”, uma das fitas mais preciosas do cinema, exibida pela primeira em vez 1896 e com apenas 50 segundos de duração.
Ao todo, foram precisas 72 mil sequências de ADN, com 200 bases (letras) cada, para guardar todos estes arquivos.
Passadas três semanas, receberam o resultado, num “único tubo de ensaio”, que basta “guardar num frigorífico chega ou no congelador para armazenamento de longa duração”, explica Dina Zielinski. Para fazer a leitura dos dados, é preciso juntar um pouco de água, um sequenciador de ADN e depois descodificar os ficheiros originais, o que demorou à equipa um dia.
“A vantagem do ADN em relação aos dispositivos de armazenamento convencionais como discos rígidos ou cassetes é que estes se tornam obsoletos e precisam de ser substituídos a cada dez anos, na melhor das hipóteses. O ADN pode durar centenas de anos”, diz Dina Zielinski. “Pense num CD dos anos 90, provavelmente hoje já está riscado. Hoje conseguimos ler o ADN de um esqueleto com 4000 anos”, explicou o colega Yaniv Erlich à “The Scientist”. “Mas uma das coisas boas do ADN é que não está sujeito à obsoloscência digital. Pense nas cassetes de vídeos ou nos filmes de 8mm. Hoje em dia é muito difícil vê-los porque as mudanças do hardware são muito rápidas. No caso do ADN, o hardware não vai a lado nenhum. Já cá anda há 3 mil milhões de anos. Se a humanidade perder a capacidade de ler ADN, teremos problemas muito maiores do que o armazenamento de dados.”
Com a nova técnica, os investigadores conseguiram armazenar 215 petabytes de informação num grama de ADN sintético. “O seu portátil tem provavelmente um terabyte. Multiplique isso por 200 mil e toda essa informação caberia num grama de ADN.” Conseguiram um recorde de 1,8 bits por nucleótido ou base. Ou seja, cada letra guarda essa quantidade de informação.
O preço é, por agora, o problema desta revolução. Desenvolver o ADN sintético custa 7000 dólares e a tecnologia para o ler leva outros 2000 dólares.